Maschinelle Datenanalyse mittels künstlicher Intelligenz
Eine Generische Pipeline für KI-basierte Datenanalyse
von DI Dr. Alexander Maletzky
Daten werden heutzutage in Unmengen aufgezeichnet und abgespeichert. Das Ziel ist dabei oftmals ein datenbasiertes Vorhersagemodell zu erstellen, mit dem sich zukünftige Entwicklungen vorhersagen lassen. Der Weg von den Rohdaten bis zum fertigen Modell ist jedoch meist weiter als gedacht. Die RISC Software GmbH entwickelte dafür eine generische Pipeline für KI-basierte Datenanalyse.
Inhalt
- Das Problem
- Unsere Lösung: Eine Generische Pipeline
- Beispielanwendung: Sterblichkeitsvorhersage in der Intensivstation
- Weiterführende Informationen
- Autor

Das Problem
State-of-the-Art Methoden der künstlichen Intelligenz, wie z.B. neuronale Netze, benötigen qualitativ hochwertige, gut aufbereitete Daten als Input, um brauchbare Ergebnisse erzielen zu können. Die Realität hält diesen Anforderungen jedoch nicht Stand: Daten enthalten Ausreißer und fehlende Werte, oder werden in unterschiedlichen – manchmal sogar unregelmäßigen – Messfrequenzen aufgezeichnet.
Was also tun?
Um die Daten trotzdem verwenden zu können, ist eine umfangreiche Aufbereitung vonnöten. Dieser Vorgang hängt natürlich von den konkreten Daten ab, umfasst aber im Wesentlichen immer dieselben Schritte:
- Importieren der Rohdaten, z.B. aus relationalen Datenbanken,
- Validieren und Harmonisieren der Daten, und
- Imputieren („Auffüllen“) fehlender Werte.
Für das nachgelagerte Trainieren eines Vorhersagemodells sind zudem noch „organisatorische“ Schritte nötig, wie z.B. das Partitionieren in Trainings- und Testdaten. Abbildung 1 stellt den gesamten Prozess der maschinellen Datenanalyse schematisch dar.

Unsere Lösung: Eine Generische Pipeline
Im Rahmen des Projekts MC3 (https://risc-software.at/mc3/), das sich mit Datenanalyse im medizinischen Umfeld befasst, haben Expertinnen und Experten der RISC Software GmbH eine generische Datenpipeline entwickelt. Damit lässt sich ein großer Teil des Datenanalyseprozesses abbilden – insbesondere die zuvor erwähnte Datenaufbereitung ist integraler Bestandteil. Zusätzlich stellt das System eine einheitliche Schnittstelle für beliebige Machine-Learning Algorithmen bzw. Modellklassen bereit, sodass das Trainieren, Anwenden und Analysieren eines Vorhersagemodells ebenfalls über die Pipeline abgebildet werden kann.
Ein spezieller Fokus liegt auch auf dem immer wichtiger werdenden Thema der Explainable AI – der erklärbaren künstlichen Intelligenz. So kann so gut wie jede State-of-the-Art Erklärungsmethode, von Layerwise Relevance Propagation bis zu Shapley Values, über eine einfache Schnittstelle integriert werden, um Modellvorhersagen für den Menschen nachvollziehbar zu machen. Die Pipeline ist so umgesetzt, dass sie möglichst wiederverwendbar ist. Sie ist modular aufgebaut, das heißt, einzelne Komponenten können beliebig kombiniert, hinzugefügt und entfernt werden. Außerdem können Endanwender die einzelnen Schritte ganz einfach konfigurieren, wie z.B. Validierungsregeln, Imputationsstrategien etc. spezifizieren. Selbst Grid Search zum Explorieren des Parameterraums ist problemlos möglich. Die Anwendbarkeit der Pipeline ist somit nicht auf medizinische Daten beschränkt.
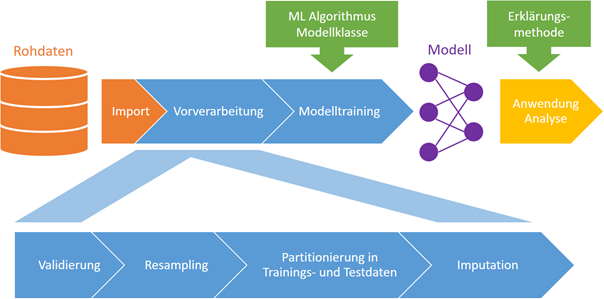
Abbildung 1. Schematische Darstellung der maschinellen Datenanalyse mittels künstlicher Intelligenz. Die wiederverwendbare Datenpipeline umfasst die in Blau dargestellten Komponenten, und unterstützt den Datenimport und die Anwendung/Analyse des trainierten Modells.
Beispielanwendung: Sterblichkeitsvorhersage in der Intensivstation
Forscherinnen und Forscher der RISC Software GmbH haben die Pipeline exemplarisch auf die öffentlich zugängliche MIMIC-III Datenbank angewendet, die in der Literatur oft als Benchmark-Datensatz im Bereich der (intensiv)medizinischen Datenanalyse verwendet wird. Ziel war es, die Sterbewahrscheinlichkeit eines Patienten in der Intensivstation anhand der ersten zwölf Stunden nach Aufnahme vorherzusagen. Dank der entwickelten Pipeline ließ sich fast der gesamte Datenanalyse- und Modellerstellungsprozess auf ein paar simple Parameterkonfigurationen beschränken. Das erzielte Ergebnis muss den Vergleich mit aktuellen wissenschaftlichen Publikationen zu diesem Thema nicht scheuen.
Die Projekte der Abteilung Medizin-Informatik werden aus Mitteln des Strategischen Wirtschafts- und Forschungsprogrammes „Innovatives OÖ 2020“ vom Land OÖ gefördert.
Weiterführende Informationen
Technologiestack: Python 3, mit den einschlägigen add-on Packages (Pandas, Plotly, scikit-learn, etc.).
MC³: Medical Cognitive Computing Center, gemeinsames Forschungsprojekt von Kepler Universitätsklinikum Linz / MedCampus III, Johannes Kepler Universität Linz / Institut für Machine Learning, und RISC Software GmbH / Abteilung Medizininformatik.
MIMIC-III Datenbank: Medical Information Mart für Intensive Care III, öffentlicher Datensatz von über 58.000 Intensivpatienten aus einem Krankenhaus in Boston, MA; https://mimic.physionet.org/
Kontakt
Autor

DI Dr. Alexander Maletzky
Researcher & Developer Unit Medical Informatics